EVRİMCİLERİN FİLOGENETİK SOYAĞAÇLARI ARASINDAKİ TUTARSIZLIKLAR
Prof. Dr. Ali Osman BELDÜZ
Karadeniz Teknik Üniv., Fen Fakültesi, Biyoloji Bölümü, Trabzon.
[email protected]
Fosiller, hayvanların tek ortak bir atadan evrimleştiğini göstermeyince, evrimciler, DNA dizilerine umut bağladılar. Moleküler verilerle çizilen ağaçların morfolojik ağaçlarla uyum gösteren, muazzam makro evrim ağaçlarını meydana getireceğini ileri sürdüler. Evrim ağaçları, “İki türün nükleotit dizileri birbirlerine ne kadar çok benziyorsa, o türler birbirlerine o kadar yakın akraba türlerdir” varsayımı esasına dayanılarak çizilir. Bu varsayım, esasen, teorinin ana özelliğinin -evrensel ortak ata fikrinin- açık ve net ifadesidir. Bununla birlikte, farklı türler arasındaki genetik benzerliklerin mutlaka ortak atadan kaynaklandığını iddia etmenin sadece bir varsayımın olduğunu bilmek önemlidir.
Bu varsayım doğru ise, farklı dizileri kullanarak teşkil edilen ağaçlar, farklı genler veya diziler arasında oldukça tutarlı bir model ve benzerlik ortaya koymalıdır. DNA dizileri ile muazzam bir "Hayat ağacı" inşa etme çabalarının beklentileri karşılamamış, bir gen hayat ağacının bir versiyonunu verirken, diğer bir gen ise, ağacın son derece farklı ve çelişkili bir versiyonunu vermiştir. Sitokrom c, b ve mikroRNA dizileri, birbirlerinden farklı ağaçlar hâsıl etmişlerdir.
Evrimci Doolittle: "Moleküler filogenistler gerçek ağacı bulmakta başarısız oldular” sözüyle, Syvanen; "Hayat ağacını yok ettik" ifadesi ile New Scientist dergisi ise; “Uzun zamandır, kutsal kase, hayat ağacını meydana getirmekti… Fakat bugün proje menfi delillerin baskısı altında param parçadır “ beyanı ile DNA dizi verileri ile teşkil edilen ağaçlar arasındaki uyumsuzlukları, ağacın kökünden dalların tepesine kadarki çelişkileri itiraf etmişlerdir.
2005 yılında birçok gen ile yapılan bir çalışma da bu sonuçları teyit etmiştir. 2012 yılında yayınlanan bir başka makalenin ifade ettiği gibi; "Filogenetik çelişkiler yaygındır ve istisna olmaktan çıkmış, sıklıkla meydana gelen bir kural haline gelmiştir".
Filogenetik Ağaçlar Arasındaki Tutarsızlıklar
Fosiller, bütün canlıların ortak tek bir atadan geldiğini göstermeyince, evrimciler bir başka delile, DNA dizilerine, umut bağladılar. Zuckerland ve Pauling (1965) gibi araştırıcılar, morfolojik ve anatomik karakterlere dayalı ağaçlarla uyum gösterecek ağaçların, DNA dizilerinin karşılaştırılması ile teşkil edilecek moleküler esaslı filogenik ağaçlara uyum göstereceğini ileri sürerek, fosil, morfolojik ve anatomik delillerden hayal kırıklığına uğrayan evrimcileri yeni umutlarla beslediler.
Moleküler evrim ağacı teşkil etmenin altında yatan temel mantık, oldukça basittir: Öncelikle çoğu organizmada mevcut olan uygun bir gen veya genler seçilir, bu genler hizalanır ve çeşitli algoritmalar kullanılarak evrim ağaçları çizilir. İki tür arasındaki nükleotit dizisi ne kadar çok benziyor ise, o iki tür birbirine o kadar çok benzerdir. Nükleotit dizileri ne kadar çok farklı ise, o iki tür birbirlerine oldukça yakındır. Türler birbirine ne kadar yakın ise, ağaçta da birbirine o kadar yakın dalda veya aynı dalda bulunurlar. Yani moleküler sistematik, genel benzerliğin derecesinin akrabalığın derecesini yansıtması esasına dayanır[1].
Bu varsayım, evrensel ortak tek bir ata fikrinin esasıdır ve sadece bir varsayımdan ibaret olduğunu hatırdan çıkarmamak gerektir. Eğer bu varsayım doğru ise, farklı genleri kullanarak teşkil edilen ağaçlar arasında tutarlı bir benzerlik bulunmalıdır[2].
Hâlbuki bir gen bir evrim ağacı verirken, başka bir gen ağacın son derece çelişkili ve farklı başka bir versiyonunu vermektedir. Mesela, standart memeli ağacında insanlar fillere göre kemirgenlere daha yakın iken, mikroRNA dizileri ile teşkil edilen ağaçlar ise tersini göstermektedir. Yani insanlar, kemirgenlerden daha fazla fillerle akrabadır. Bu temel bir problemdir. DNA veya diğer biyolojik verileri kullanarak yapılan ağaçlar, beklentileri karşılayamamış, beklenen muazzam evrim ağacını vermemiş, yeni tutarsızlıklara, çelişkilere kapı aralamıştır. Bu durum yukarıda ifade ettiğimiz varsayımın doğru olup olmadığı konusunda haklı soruları ortaya çıkarmıştır.
Sadece RNA dizilemekten öteye, gerçek bakteri ve arke genlerinin dizilenmesinin mümkün olduğu 1990’ların başından sonra, bu problemler kendilerini gösterdi. Beklentiler, RNA dizilerinin hâsıl ettiği ağaçların aynısını DNA dizilerinin meydana getireceği yönünde idi, ama sonuçlar her zaman beklentilere uymadı. Bazen tutarlı, bazen tutarsız, farklı ağaçlar elde edildi. Mesela, RNA A türünü B türüne C türünden daha yakın akraba kılarken, DNA dizileri ile yapılan ağaçlar ise bunun tersini gösterdi[3].
Bu durum evrimci biyokimyacılardan W. Ford Doolittle'in başarısızlığı itiraf etmesine sebep oldu. W. Ford Doolittle; Moleküler filogenistlerin “Gerçek ağacı” bulmakta başarısız olduklarını, bunun sebebinin metotlarının uygun olmaması ya da yanlış genleri seçmiş olmalarından kaynaklanmış olabileceğini veya hayatın bir ağaç şeklinde gösterilmesinin uygun olmayacağı” açıklamasını yapmak durumunda kaldı[4].
New Scientist bunu şu şekilde ifade etti: [5].
Bu tutarsızlıkların, tezatların, çelişkilerin teşekkül sebebi, her ne kadar bakteriler arasında gerçekleşen horizontal gen transferine dayandırılsa da, bu tezatlar sadece bakterilerin evrim ağaçlarında değil, yüksek yapılı canlıların evrim ağaçları arasında da görülmektedir. Moleküler evrim sistematiğinin öncüsü Carl Woose (1998), bu çelişkilerin evrensel evrim ağacının kökünden dallarına kadar her yerde, taksonların içinde veya aralarında görülebileceğini itiraf etmiştir.
2009 yılında New Scientist dergisinde Graham Lawton (2009) tarafından neşredilen "Why Darwin was wrong about the tree of life” başlıklı makalede de bilim adamları; “Evrimin tamamen ağaç benzeri olmadığı” önerisine yer vermiştir.
Ayrıca makale Michael Syvanen’in 2000 gen kullanarak farklı hayvan grupları arasında akrabalıkları gösteren bir ağaç hâsıl etme girişiminin sonuçlarından bahsedilmiştir. Bu teşebbüsün başarısız olduğu, genlerin karışık sinyaller gönderdiği, farklı genlerin farklı evrim ağaçları hâsıl ettiği, genlerin yarısının bir evrim hikayesi, diğer yarısının başka bir evrim hikayesi teşkil ettiği ifade edilmiştir. Neticede Syvanen’in; “Evrim ağacını yok ettik” itirafına yer verilmiştir. Literatürde benzeri problemlerden sıkça bahsedilmektedir[6].
Trends in Ecology and Evolution’da 2009’da yayınlanan bir makale, bu çelişkilerin sebebini, bu kadar büyük veriyi teşkil eden genomun farklı genlerinin farklı genealojik (şecere) geçmişlere sahip olma ihtimalinin olduğunu ileri sürüyor[7]. Benzeri bir çelişki, Genome Research’da “farklı proteinlerin farklı filogenetik ağaçlar ürettiğini” gösteren bir makalede de ifade ediliyor[8].
2012 yılında Nature dergisinde mikroRNA dizileri kullanılarak yapılan evrim ağaçları ile ilgili bir makale ise, mikroRNA’ların geleneksel hayvan evrim ağaçlarını çöpe attığını, mikroRNA’ların geleneksel hayvan evrim ağaçlarını destekleyecek sonuçlar vermediğini, mikroRNA’ların memeliler için tamamen farklı diyagramlar hâsıl ettiğini iddia etmiştir. mikroRNA dizileri ile yapılan evrim ağaçları herkesin beklediğinden, evrimcilerin hayalinden farklı ağaçlar üretmekte, insanları kemirgenlere göre fillere daha yakın göstermektedir (Şekil 1)[9].
Morfolojik veriler ile moleküler verilere dayalı ağaçlar arasındaki uyuşmazlıklar yaygın bir durumdur. Anatomik ve morfolojik karakterlere dayalı ağaçlarla DNA’ya dayalı ağaçlar arasındaki çelişkileri karşılaştıran tek makale olan 2012 tarihli çalışmada, “hem karakter hem de veri setleri hızla artarken bu çelişkilerin de yaygınlaştığı” ifade edilmiştir[10].
Ders kitaplarında genelde morfolojik verilere dayalı geleneksel evrim ağaçları ile uyum gösteren sitokrom c enzimine dayalı hayvanlara ait bir ağaç misal gösterilmektedir (Şekil 2)[11]. Bununla birlikte, ders kitapları, nadiren farklı bir enzime (sitokrom b) dayalı ağacın, morfolojik verilere dayalı evrim ağacı ile uyumlu olmadığından bahseder.

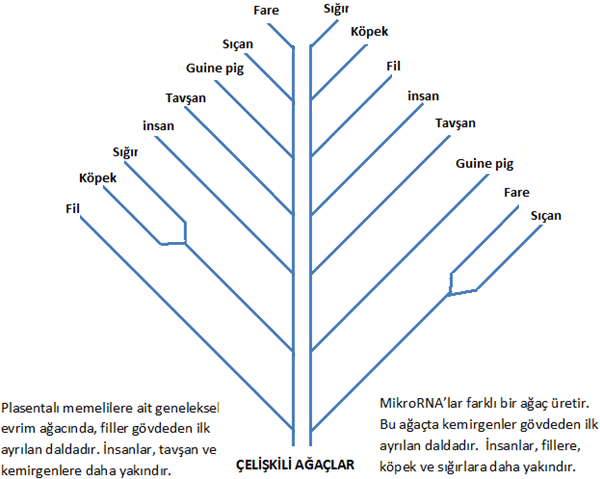
Şekil 1. Geleneksel evrim ağacı ile mikroRNA dizileri kullanılarak elde edilen evrim ağaçları arasındaki tezatlar. Dolgin, 2012’den modifiye edilmiştir.


Şekil 2. Sitokrom c’ye dayalı evrim ağacı[12].
Hâlbuki sitokrom b, omurgalılarda, muhtemelen en sık dizilenen ve soyağaçlarının teşkilinde sıkça kullanılan genlerden biridir. Sitokrom b dizilerine dayalı ağaçlar, ağaç meydana getirme algoritmasına bakılmaksızın sitokrom c’den farklı bir evrim ağacı hâsıl eder (Şekil 3)[13].

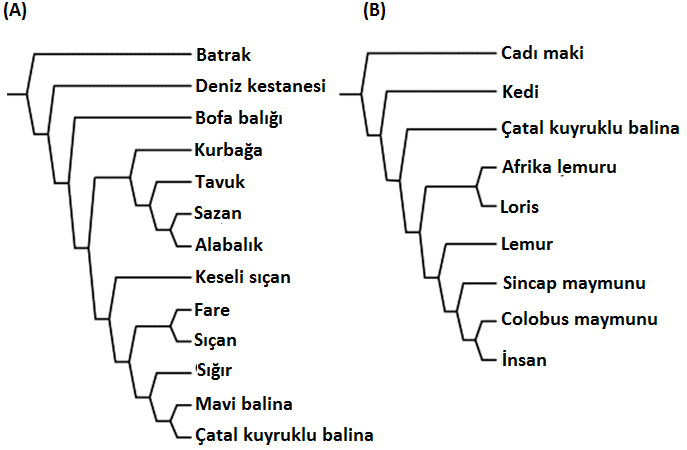
Şekil 3. Moleküler dizilere dayalı beklenmedik evrim ağaçlarları. (a) Protein kodlayan mitokondri genomunun tamamı ile çizilen omurgalı ağacı; deniz kestanelerini omurgalılar arasına yerleştiriyor, kurbağalar, kuşlar ve balıklar ayrı bir klad yapıyor. (b) Sitokrom b gen dizileri ile çizilen ağaç; Tarsier’ler (cadı makiler) diğer primatlara kedi ve balinalardan daha uzaktır. Lee, 1999’dan modifiye edilmiştir.
2000 yılında Nature dergisinde Gura (2000) tarafından yazılan bir makalede ise, moleküler ve morfolojik verilerin farklı moleküler ağaçlar vererek tezat teşkil ettiklerinden bahsediliyor. Moleküler ve morfolojik veriler, organizmaları ağaçlarda farklı yerlere yerleştirerek, evrim ağaçları içindeki çelişkilere tekrar parmak basıyor (Şekil 4). 

Şekil 4. Neredeyiz? Primatlar arasındaki morfolojik ve moleküler ilişkileri gösteren ağaçlar. Soldaki dendogram moleküler verilere, sağdaki dendogram morfolojik verilere dayanılarak çizilmiştir[14].
Moleküler evrim ağaçları arasındaki çelişkilere ait bir başka misal, mitokondri genomundaki iki gene dayalı ağaçların arasındaki çelişkidir. Hem COB (sitokrom b) hem’de COII (sitokrom c oksidaz altbirim II) genlerine dayalı evrim ağaçları arasındaki çelişki aşağıdaki şekilde görülebilir. Sitokrom b’ye göre insan kemirgenlere yakın akraba iken, sitokrom c oksidaz altbirim II’ye göre ise bir primat ile akraba oluyor (Şekil 5)[15].


Şekil 5. Plasentalı memelilerin maksimum parsimoniye dayalı filogenetik ilişkilerini gösteren ağaçlar. (A) Mitokondri sitokrom b geni, (B) Mitokondri sitokrom c oksidaz altbirim II geni[16].
Dikkat çekici bir şekilde, Trends in Ecology and Evolution’deki farklı bir makalede şöyle bir sonuca varılıyor:
“Memeli ordoları üzerine yapılmış, çokça benimsenmiş hem moleküler ve hem de morfolojik filogeniler, memeli ağacını çözümsüz bir çalılığa indirger”[17].
Böyle çelişkiler sebebiyle, Nature’deki önemli bir derleme makale; “Evrim savaşlarına” sebep olan “moleküler ve morfolojik ağaçlar arasındaki ihtilafı” analiz etmiştir. Biyolojik molekülleri araştırarak teşkil edilen evrim ağaçlarının, morfolojiye dayanarak yapılanlara benzemediği belirtilmiştir[18].
Dávalos ve arkadaşlarının (2012) bir makalesinde ifade ettiği gibi, filogenetik çatışma (uyuşmazlık) yaygındır ve istisna olmaktan çok, sıklıkla görülen bir kuraldır.
Bütün bunların sonunda, DNA dizi verilerinin güzel düzgün bir evrim ağacı hâsıl edeceği ile ilgili umutlar ile birlikte Neo-Darwinci teorinin anahtar öngörüsü/beklentisi de yıkılmış durumdadır.
[1] Schwartz Jeffrey H., Maresca Bruno. Do Molecular Clocks Run at All? A Critique of Molecular Systematics," Biological Theory. 2006,1(4):357-371.
[2] Schwartz ve Maresca, 2006.
[3] Lawton G. Why Darwin was wrong about the tree of life. New Scientist. 2009, 2692: 34–39.
[4] Doolittle W. Ford. Phylogenetic Classification and the Universal Tree. Science. 1999, 284:2124-2128.
[5] Lawton, 2009.
[6] Lawton, 2009.
[7] Degnan James H. and Rosenberg Noah A. Gene tree discordance, phylogenetic inference and the multispecies coalescent. Trends in Ecology and Evolution. 2009, 24:332-340.
[8] Mushegian Arcady R., Garey James R., Martin Jason and Liu Leo X. Large-Scale Taxonomic Profiling of Eukaryotic Model Organisms: A Comparison of Orthologous Proteins Encoded by the Human, Fly, Nematode, and Yeast Genomes. Genome Research. 1998, 8:590-598.
[9] Dolgin Elie. Rewriting Evolution. Nature. 2012, 486:460-462.
[10] Dávalos Liliana M., Cirranello Andrea L., Geisler Jonathan H., and Simmons Nancy B. Understanding phylogenetic incongruence:lessons from phyllostomid bats. Biological Reviews of the Cambridge Philosophical Society. 2012, 87:991-1024.
[11] Mader Sylvia S., Isaacson Jeffrey A., Lyle-Ippolito Kimberly G., Storfer Andrew T. Inquiry Into Life, 13th ed. McGraw-Hill Book Co., 2011, pp. 550. New York, USA.
[12] Mader Sylvia S., Biology, 9th Edition, Dubuqua, IA: McGraw-Hill Book Co. 2007. New York, USA. Sylvia S.Mader, 2007’den modifiye edilmiştir.
[13] Lee Michael S. Y. Molecular Phylogenies Become Functional. Trends in Ecology and Evolution. 1999, 14:177.
[14] Gura Trisha. Bones, Molecules or Both? Nature. 2000, 406:230-233. Gura, 2000’den modifiye edilmiştir.
[15] Honeycutt, Rodney L.; Adkins, Ronald M.; Nedbal, Michael A.; Janecek, Laura L. Mammalian mitochondrial DNA evolution: A comparison of the cytochrome b and cytochrome c oxidase II genes. Journal of Molecular Evolution. 1995, 40:260-272.
[16] Honeycutt, 1995’den modifiye edilmiştir.
[17] De Jong W. W. Molecules remodel the mammalian tree. Trends in Ecology and Evolution, 1998. 13(7):270-274.
[18] Rokas Antonis & Carroll Sean B. Bushes in the Tree of Life. PLoS Biology. 2006, 4(11): 1899-1904.
BENZER SORULAR
- Filogenetik ağaç, evrim soy ağacı; canlıları uzak mı tutuyor?
- Moleküler Filogeni Hikâyesi (Bilim 'Yaratılış' Diyor-23)
- EVRİMİN DELİLİ OLARAK İLERİYE SÜRÜLEN ARA (GEÇİŞ) FORMLAR
- EVRİM TEORİSİ’NİN ÇIKMAZLARI
- EVRİM GÖRÜŞÜNDE TEMEL YANILGILAR: 1 BİLİMSELLİK, İNANÇ VE TARAFSIZLIK KARMAŞASI
- PSÖDOGENLERİN EVRİM VE YARATILIŞ AÇISINDAN İNCELENMESİ
- Ulaşılamayan Atalarımız
- Ulaşılamayan Atalarımız
- KUR’ANIN IŞIĞINDA TAHIL BİTKİLERİ
- “Evrim teorisi ispatlandı.” şeklinde yapılan açıklamaların doğruluk payı var mıdır?

